miRNA的靶基因预测详细教程
目录
最近发现很多人对预测miRNA的靶基因感兴趣,因此分享一下这方面的知识。通过生信算法预测miRNA靶基因的网站有很多,下面例举3个常见网站,并给出详细使用教程。
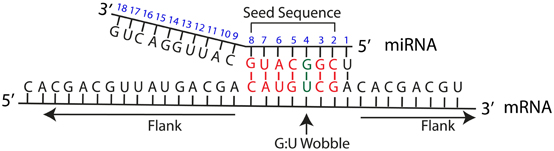
一般来说,预测网站的假阳性会比较高,我们可以同时用这些网站进行预测,然后取交集来提高准确率。下面以miR-320为例进行说明。
miRTarBase
以hsa-miR-320a为例,查询后的结果如下图所示:
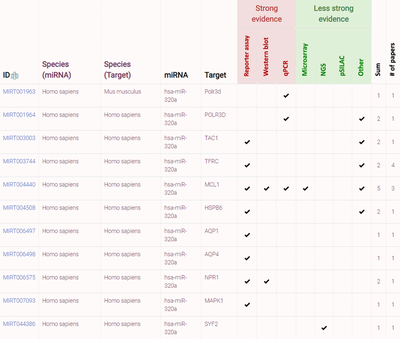
其中值得注意的是:所有的相互作用对来源于6种证据,有reporter assay, western blot, qPCR, Microarray, NGS, PSILAC。前面三种证据的可靠较强。
miRDB
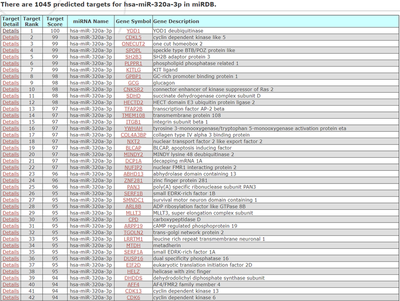
miRDB的可靠性则通过Target Score来体现,从高到低进行排序。点击相对应的Details可以查看相匹配的位点。
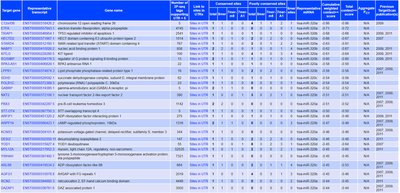
TargetScan
TargetScan 则通过搜索mRNA的保守 8mer, 7mer, 和6mer 位点来与miRNA相匹配。然后按一个总的得分进行排序。
批量分析
除了单个miRNA输入查询外,这3个网站同时也提供了数据下载服务。我们可以把miRNA-mRNA相互作用的关系对下载下来,然后用R语言处理一下,就可以批量分析miRNA的靶基因了。
下面是针对人类的预测分析:
rm(list=ls());options(stringsAsFactors=F)
library(stringr)
library(biomaRt)
miRNAs="miR-320a";species="hsa"
source=paste0(species,'-',miRNAs)
miRTarBase = read.csv("miRTarBase_MTI.csv",head=T)
miRDB = read.table("miRDB_v5.0_prediction_result.txt",head=F)
targetscan = read.table("Predicted_Targets_Context_Scores.default_predictions.txt",head=T,sep="\t")
## miRTarBase
miRTarBase_mRNA = miRTarBase[str_detect(miRTarBase$miRNA,paste0('^',source,'$|^',source,'-[35]p')),]
## miRDB
miRDB_mRNA = miRDB[str_detect(miRDB$V1,paste0('^',source,'$|^',source,'-[35]p')),]
ensembl = useEnsembl(biomart="ensembl", dataset="hsapiens_gene_ensembl")
miRDBgene = getBM(attributes = c( 'refseq_mrna',"hgnc_symbol"), filters = 'refseq_mrna', values = unique(miRDB_mRNA$V2), mart = ensembl)
miRDBgene = miRDBgene[miRDBgene$hgnc_symbol!="",]
miRDB_mRNA = merge(miRDB_mRNA,miRDBgene,by.x="V2",by.y="refseq_mrna",sort=F)
## TargetScan
targetscan_mRNA = targetscan[str_detect(targetscan$miRNA,paste0('^',source,'$|^',source,'-[35]p')),]
## merge
res = merge(miRTarBase_mRNA,targetscan_mRNA,by.x="Target.Gene",by.y="Gene.Symbol")
res = merge(res,miRDB_mRNA,by.x="Target.Gene",by.y="hgnc_symbol")
write.csv(res,file="miRNAtargetGene.csv",row.names = F,col.names = F)
这个代码还不完善,之后有机会把它们做成一个R软件包,更方便使用。
请问后续有做R包吗?谢谢~
您好,阅读了您的文章对我的学习非常有帮助。不过有些不明白的地方还想请教您,请问targetscan数据库这行代码中【targetscan = read.table(“Predicted_Targets_Context_Scores.default_predictions.txt”,head=T,sep=”\t”)】读入的原数据是在哪里下载的呢?
@Anonymous , 好的,非常感谢,我去学习以下 
v1.5.2